Cell Ranger
上一节我们学习了10X Genomics的测序原理,这节我们来看看如何使用官方推荐的软件cell ranger来分析数据。Cell Ranger是由10x Genomics公司提供的一套用于处理单细胞RNA测序数据的工具。官网对其进行了详细的介绍和使用说明。可以通过10x Genomics Cloud Analysis使用这个软件或者将其安装到本地。
安装
CellRanger必须在Linux系统下运行。首先去下载Linux版本对应的软件安装包,下载之前需要先提交许可证相关信息。然后登录自己的服务器并使用mkdir 和cd等命令建立并进入到软件安装目录。使用下面的命令下载软件。
curl -o cellranger-6.1.2.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-6.1.2.tar.gz?Expires=1641366506&Policy=eyJTdGF0ZW1lbnQiOlt7IlJlc291cmNlIjoiaHR0cHM6Ly9jZi4xMHhnZW5vbWljcy5jb20vcmVsZWFzZXMvY2VsbC1leHAvY2VsbHJhbmdlci02LjEuMi50YXIuZ3oiLCJDb25kaXRpb24iOnsiRGF0ZUxlc3NUaGFuIjp7IkFXUzpFcG9jaFRpbWUiOjE2NDEzNjY1MDZ9fX1dfQ__&Signature=kaV8~ZabHhyDykUhbN~F78PDQfNZ64IamgsGc1nOSghFKPr0fbZ3WJk-2eWYh7IEt-KupenYP89W1zHi4lrxF~ZBbuP4NTaKEAa-G6ILJoX-VdyFnktkXFYDHgzEJ8ABq-NM6RWn20WD3a9BITNHTIWPtxjM-NaXAuR5uc5PuAEgjSDaQ2QBAQr~1q4aSM-~vJt~ia5e8acTz9RlM24EluLqfO59VCtAorP-5iJRwvLw9DjfrTlDtWfy3M2LSXp5OGmVJH1WUQReLK~0iZX2e8~vrHlAYpuxMa0Lgil6oHQ5s6vc~Dod3Aqpjb9sM~wuVo80zi4EqJ5nq0LU8SNbiQ__&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA"如果系统没有curl命令,可以使用wget命令,复制如下命令行:
wget -O cellranger-6.1.2.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-6.1.2.tar.gz?Expires=1641366506&Policy=eyJTdGF0ZW1lbnQiOlt7IlJlc291cmNlIjoiaHR0cHM6Ly9jZi4xMHhnZW5vbWljcy5jb20vcmVsZWFzZXMvY2VsbC1leHAvY2VsbHJhbmdlci02LjEuMi50YXIuZ3oiLCJDb25kaXRpb24iOnsiRGF0ZUxlc3NUaGFuIjp7IkFXUzpFcG9jaFRpbWUiOjE2NDEzNjY1MDZ9fX1dfQ__&Signature=kaV8~ZabHhyDykUhbN~F78PDQfNZ64IamgsGc1nOSghFKPr0fbZ3WJk-2eWYh7IEt-KupenYP89W1zHi4lrxF~ZBbuP4NTaKEAa-G6ILJoX-VdyFnktkXFYDHgzEJ8ABq-NM6RWn20WD3a9BITNHTIWPtxjM-NaXAuR5uc5PuAEgjSDaQ2QBAQr~1q4aSM-~vJt~ia5e8acTz9RlM24EluLqfO59VCtAorP-5iJRwvLw9DjfrTlDtWfy3M2LSXp5OGmVJH1WUQReLK~0iZX2e8~vrHlAYpuxMa0Lgil6oHQ5s6vc~Dod3Aqpjb9sM~wuVo80zi4EqJ5nq0LU8SNbiQ__&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA"注意:如果上述链接失效,可以去官网复制最新命令
下载界面如下所示:
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 767M 100 767M 0 0 124M 0 0:00:06 0:00:06 --:--:-- 131M下载完成之后,使用ls命令查看下载的文件
$ ls #查看下载的文件
cellranger-6.1.2.tar.gz
$ tar -zxvf cellranger-6.1.2.tar.gz #解压文件,如果是.tar.xz后缀,使用tar -xvf命令
cellranger-6.1.2/
cellranger-6.1.2/.env.json
cellranger-6.1.2/.version
cellranger-6.1.2/LICENSE
cellranger-6.1.2/builtwith.json
...
$ cd cellranger-6.1.2
$ pwd #查看绝对路径
/path/to/your/workdir/cellranger-6.1.2
$ export PATH=/mnt/home/user.name/yard/apps/cellranger-6.1.2:$PATH #添加路径到系统环境中
$ cellranger #查看是否可以正常使用
USAGE:
cellranger
FLAGS:
-h, --help Prints help information
-V, --version Prints version information
SUBCOMMANDS:
count Count gene expression (targeted or whole-
transcriptome) and/or feature barcode reads from a
single sample and GEM well
multi Analyze multiplexed data or combined gene
expression/immune profiling/feature barcode data
...
使用
cellranger提供了五个分析模块用于分析单细胞转录组相关产品
1.cellranger mkfastq:
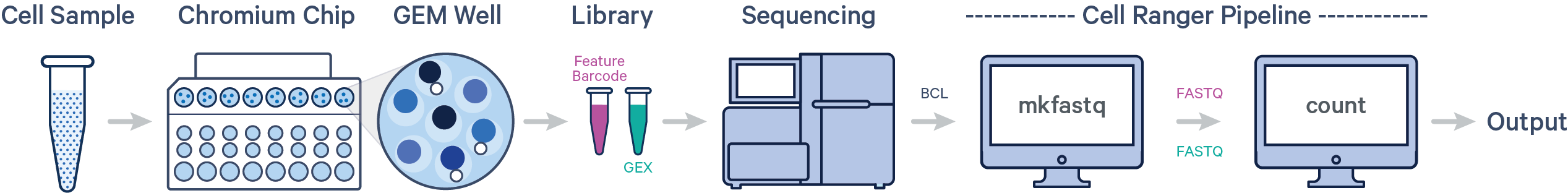
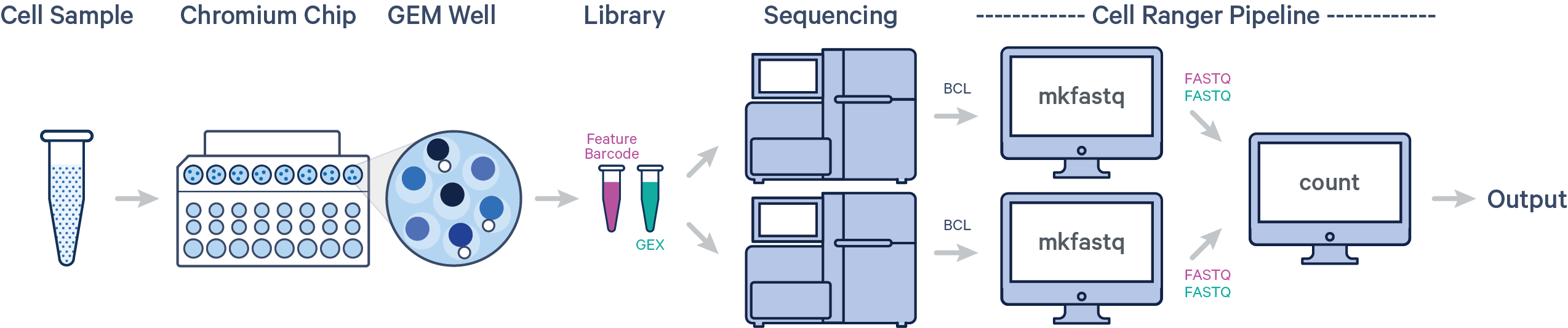
调用bcl2fastq软件将illumina测序仪得到的BCL( raw base call )格式文件拆分为fastq文件。工作流如图所示:

一次上机测序两个不同的文库,执行一次cellranger mkfastq

一次上机测序两个不同的文库,分别执行一次cellranger mkfastq。
使用方法
$ cellranger mkfastq --id=tiny-bcl \
--run=/path/to/tiny_bcl \
--csv=cellranger-tiny-bcl-simple-1.2.0.csv参数说明
| Parameter | Function |
|---|---|
--run |
必选;BCL文件所在目录 |
--id |
可选;mkfastq将会生成相应目录,不接受绝对路径 |
--samplesheet --sample-sheet |
可选. 样本名字、index等信息; [Data] Lane,Sample_ID,index,index2 1,test_sample,TGGTCCCAAG,ACGCCAGAGG |
--csv--simple-csv |
可选,类似上面参数 |
| … | … |
--output-dir |
指定FASTQ输出目录 |
--localcores |
运行的最大CPU数 |
--localmem |
运行的最大内存数 |
2.cellranger count:
分析cellranger mkfastq或者其他方式得到fastq格式的文件。这个模块包括比对、过滤、barcoding计数以及UMI计数。可以生成barcode-UMI信息,进行聚类及基因表达分析。不过我们通常用到表达信息,使用其他方式进行聚类及其他单细胞相关分析。
在分析count之前需要先下载参考基因组:
$ wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2020-A.tar.gz" #人
$ wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-mm10-2020-A.tar.gz" #鼠或者参照教程使用cellranger mkref自己构建参考基因组
$ cellranger count --id=sample345 \
--transcriptome=/opt/refdata-gex-GRCh38-2020-A \
--fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \
--sample=mysample \
--localcores=8 \
--localmem=64输出结果包括以下信息:
Outputs:
- Run summary HTML: /outdir/outs/web_summary.html
- Run summary CSV: /opt/sample345/outs/metrics_summary.csv
- BAM: /opt/sample345/outs/possorted_genome_bam.bam
- BAM index: /opt/sample345/outs/possorted_genome_bam.bam.bai
- Filtered feature-barcode matrices MEX: /opt/sample345/outs/filtered_feature_bc_matrix
- Filtered feature-barcode matrices HDF5: /opt/sample345/outs/filtered_feature_bc_matrix.h5
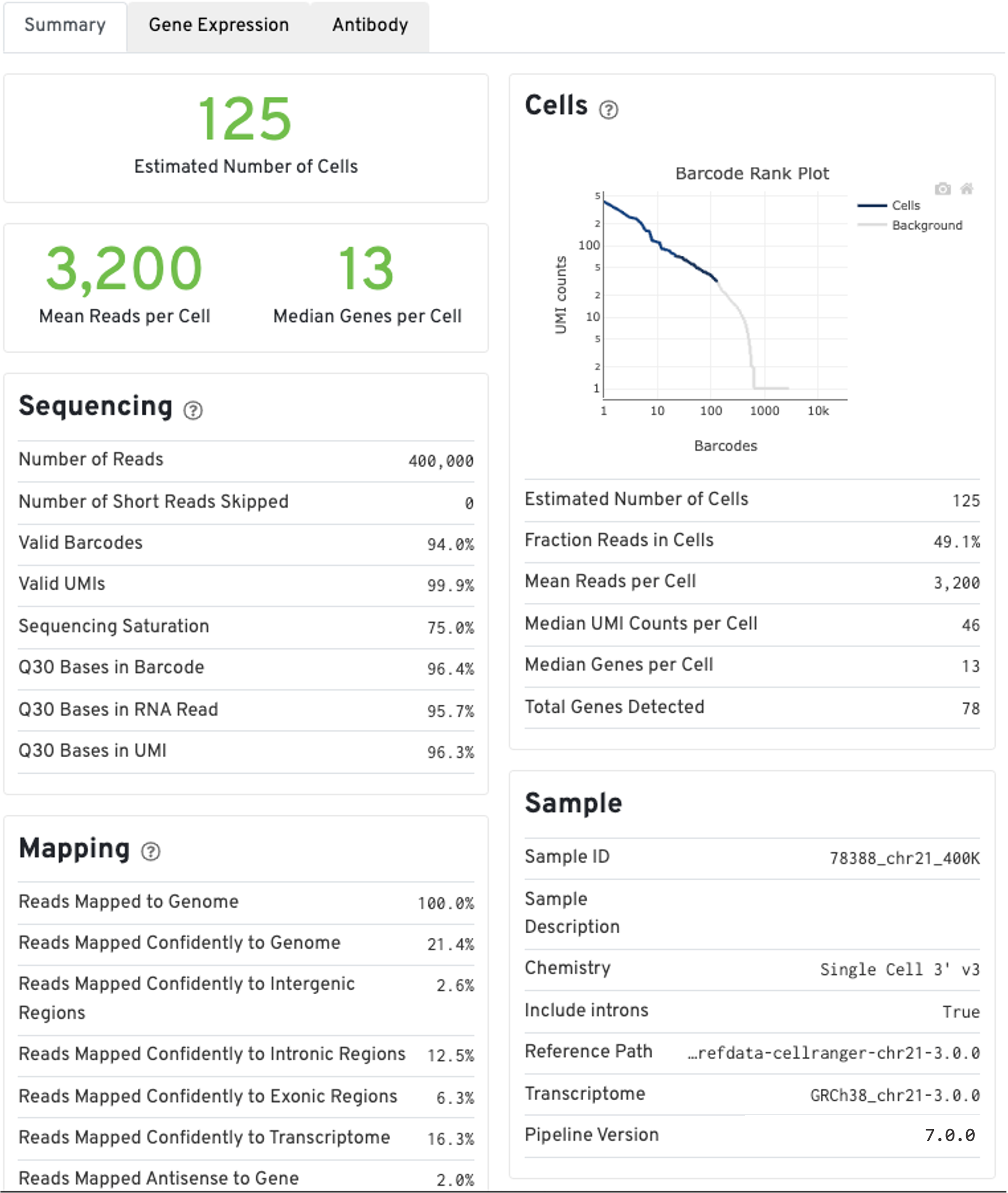
......count分析完成之后可以使用浏览器打开web_summary.html文件查看报告,网页版报告主要包括以下几个方面的内容:
Summary包括鉴定到的细胞数、reads数中值、基因数中值、比对率、饱和度等基本信息
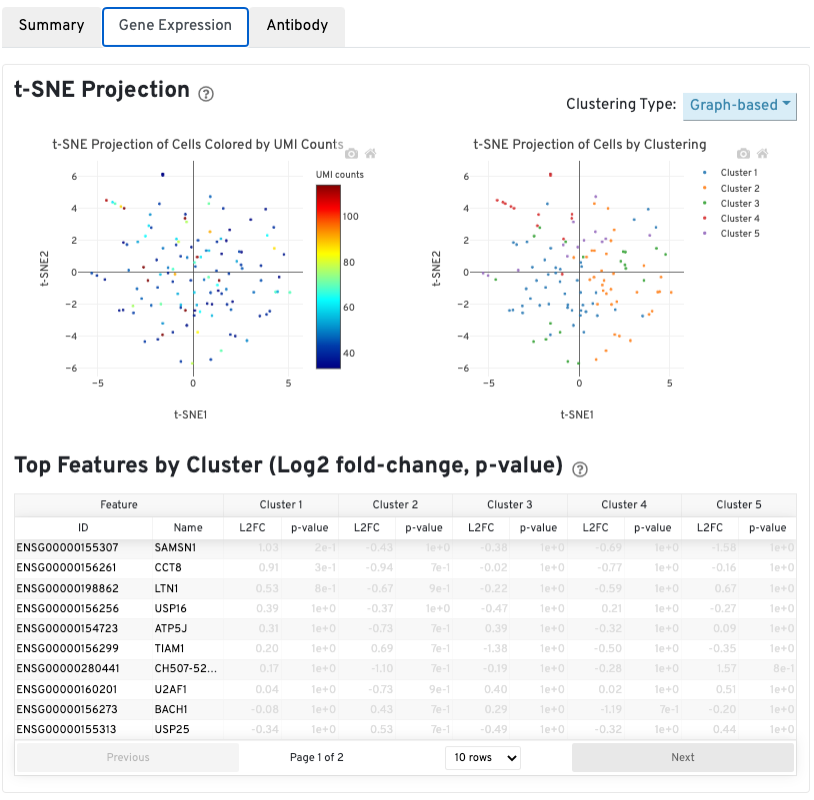
Gene Expression View包括T-SNE降维结果、初步的聚类及每个cluster的高表达基因。
建议结果部分我们重点关注Summary,来判断文库构建及测序是否合格。
3.cellranger multi:
用于分析细胞混合或者固定RNA数据,和count类似,multi也包括比对、过滤、barcoding计数以及UMI计数。可以生成barcode-UMI信息,进行聚类及基因表达分析等。
4.cellranger aggr:
用于整合count或者multi的结果。
5.cellranger reanalyze:
对count multi aggr的结果进行重新分析,分析过程中可以调整参数以达到重新聚类的目的。
总结
不同情景下各模块的分析如下所示:
单样本单次建库单次测序:mkfastq+count
单样本单次建库多次上机:多个mkfastq+单次count
单样本多次建库单次上机:单次mkfastq+多次count+单次aggr
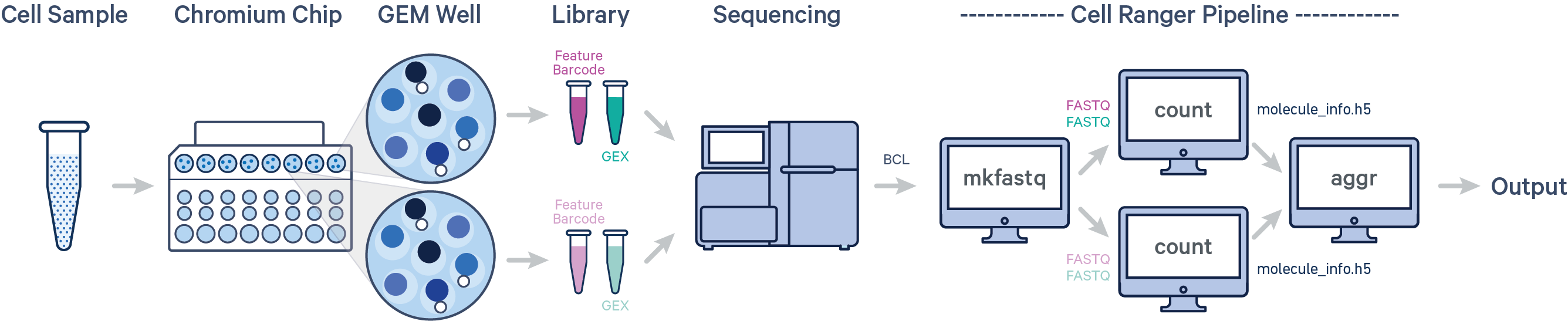
多样本多次建库单次上机:单次mkfastq+多次count+单次aggr
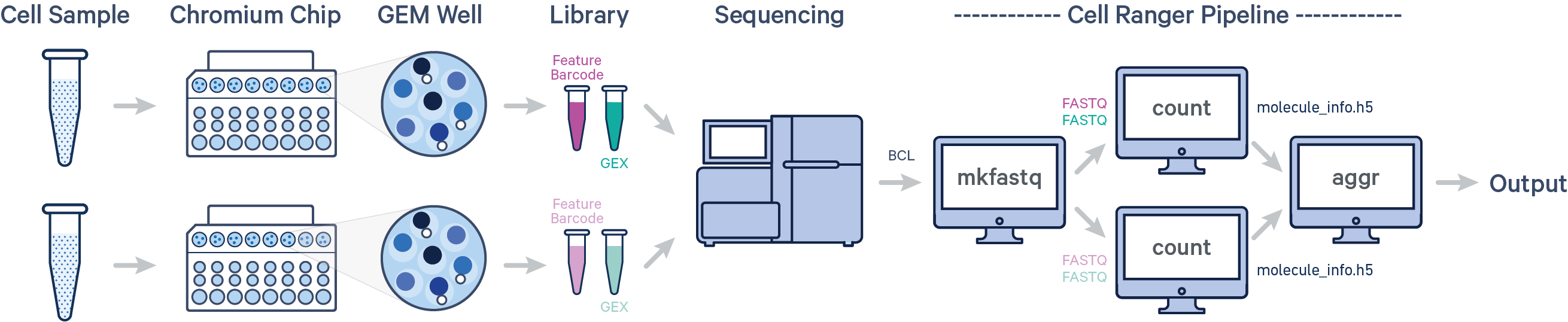
多样本单次建库单次上机:单次mkfastq+单次multi
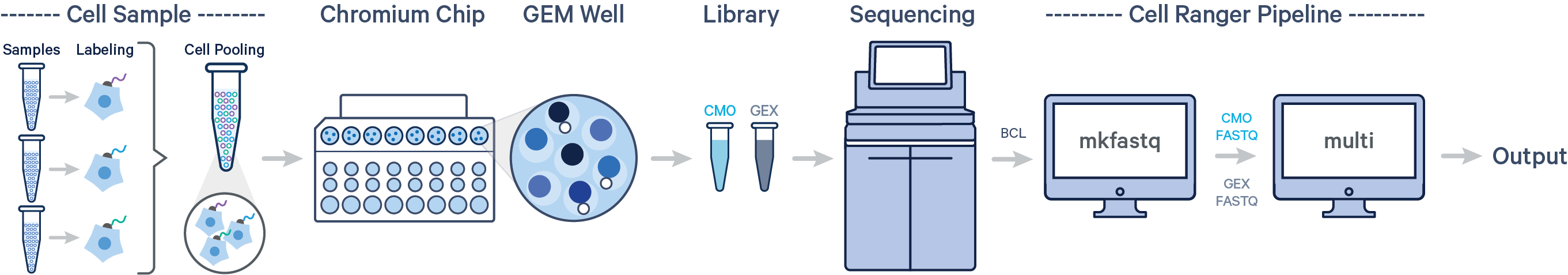
多样本单次建库单次上机:单次mkfastq+单次multi
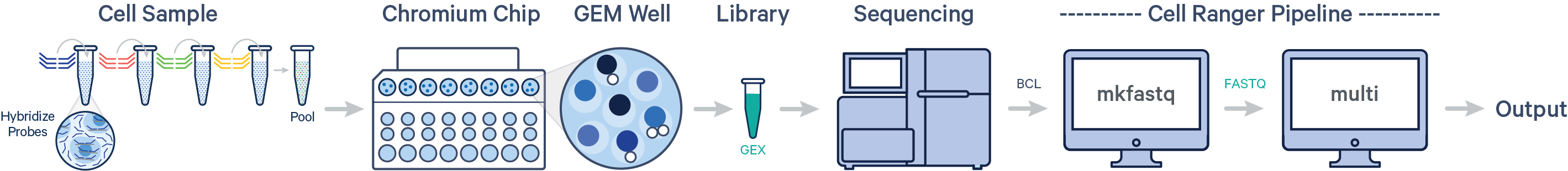
除非注明,文章均为原创,转载请以链接形式标注本文地址
本文地址:http://bioschool.cn/omics/scrna/cell-ranger/





